СТБ ISO 21571-2016
Продукция пищевая. Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов. Экстрагирование нуклеиновых кислот
Купить СТБ ISO 21571-2016 — бумажный документ с голограммой и синими печатями. подробнее
Цена на этот документ пока неизвестна. Нажмите кнопку "Купить" и сделайте заказ, и мы пришлем вам цену.
Распространяем нормативную документацию с 1999 года. Пробиваем чеки, платим налоги, принимаем к оплате все законные формы платежей без дополнительных процентов. Наши клиенты защищены Законом. ООО "ЦНТИ Нормоконтроль"
Наши цены ниже, чем в других местах, потому что мы работаем напрямую с поставщиками документов.
Способы доставки
- Срочная курьерская доставка (1-3 дня)
- Курьерская доставка (7 дней)
- Самовывоз из московского офиса
- Почта РФ
Устанавливает общие требования и специфические методы экстрагирования, очистки и количественной оценки дезоксирибонуклеиновой кислоты. Эти методы изложены в приложениях A и B.
Стандарт разработан для пищевых матриц, но может быть также применен к другим матрицам, например зерновым культурам и кормам.
Стандарт следует применять совместно с ISO 21569, ISO 21570 в части аналитических методов на основе нуклеиновых кислот, в частности качественных аналитических методов, установленных в ISO 21569, и количественных аналитических методов, установленных в ISO 21570.
- Заменяет СТБ ISO 21571-2008 «Продукты пищевые. Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов. Экстрагирование нуклеиновых кислот» ИУ ТНПА № 4-2016
Стандарт идентичен международному стандарту ISO 21571:2005
Оглавление
1 Область применения
2 Нормативные ссылки
3 Принцип метода
3.1 Общие положения
3.2 Экстрагирование ДНК
3.3 Количественная оценка содержания ДНК
4 Общие требования к лаборатории
5 Методика
5.1 Приготовление пробы для анализа
5.2 Экстрагирование и очистка ДНК
5.3 Количественная оценка экстрагированной ДНК
5.4 Стабильность экстрагированной ДНК
6 Оценка результатов
7 Протокол испытаний
Приложение A (обязательное) Методы экстрагирования ДНК
Приложение B (обязательное) Методы количественной оценки экстрагированной ДНК
Библиография
Приложение Д.А (справочное) Сведения о соответствии государственных стандартов ссылочным международным стандартам
| Дата введения | 01.01.2021 |
|---|---|
| Добавлен в базу | 01.02.2020 |
| Завершение срока действия | 01.06.2020 |
| Актуализация | 01.01.2021 |
Организации:
| 23.05.2016 | Утвержден | Госстандарт Республики Беларусь | 37 |
|---|---|---|---|
| Разработан | БелГИМ | ||
| Издан | БелГИСС | 2016 г. |
Foodstuffs. Methods of analysis for the detection of genetically modified organisms and derived products. Nucleic acid extraction
- ГОСТ ИСО 21569-2009 Продукты пищевые. Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов. Методы качественного обнаружения на основе анализа нуклеиновых кислот
- ГОСТ ИСО 21570-2009 Продукты пищевые. Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов. Количественные методы, основанные на нуклеиновой кислоте
- СТБ ISO 24276-2012 Продукция пищевая. Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов. Общие требования и определения
Чтобы бесплатно скачать этот документ в формате PDF, поддержите наш сайт и нажмите кнопку:

стр. 1

стр. 2

стр. 3

стр. 4

стр. 5

стр. 6

стр. 7

стр. 8

стр. 9

стр. 10

стр. 11

стр. 12

стр. 13

стр. 14

стр. 15

стр. 16

стр. 17

стр. 18
стр. 19

стр. 20

стр. 21

стр. 22

стр. 23

стр. 24

стр. 25

стр. 26
стр. 27

стр. 28

стр. 29

стр. 30
государственный стандарт
РЕСПУБЛИКИ БЕЛАРУСЬ
ПРОДУКЦИЯ ПИЩЕВАЯ
Методы анализа для обнаружения генетически модифицированных организмов и производных продуктов
Экстрагирование нуклеиновых кислот
ПРАДУКЦЫЯ ХАРЧОВАЯ
Метады анал1зу для выяулення генетычна мадыф1каваных аргашзмау i вытворных прадуктау
Экстрагаванне нуклешавых мслот
(ISO 21571:2005, ЮТ)
Издание официальное
Госстандарт
Минск
GS
УДК 664:543.062:547.963.32(083.74)(476) МКС 67.050 КП 03 ЮТ
Ключевые слова: продукция пищевая, дезоксирибонуклеиновая кислота (ДНК), генетически мо-дифицированные организмы (ГМО), количественная оценка, валидация_
Предисловие
Цели, основные принципы, положения по государственному регулированию и управлению в области технического нормирования и стандартизации установлены Законом Республики Беларусь «О техническом нормировании и стандартизации».
1 ПОДГОТОВЛЕН республиканским унитарным предприятием «Белорусский государственный институт метрологии» (БелГИМ)
ВНЕСЕН Госстандартом Республики Беларусь
2 УТВЕРЖДЕН И ВВЕДЕН В ДЕЙСТВИЕ постановлением Госстандарта Республики Беларусь от 23 мая 2016 г. №37
3 Настоящий стандарт идентичен международному стандарту ISO 21571:2005 Foodstuffs -Methods of analysis for the detection of genetically modified organisms and derived products - Nucleic acid extraction (Продукты пищевые. Методы анализа для обнаружения генетически модифицированных организмов и их производных. Извлечение нуклеиновой кислоты).
Международный стандарт разработан техническим комитетом по стандартизации СЕИЯС 275 «Анализ пищевой продукции. Горизонтальные методы» Европейского комитета по стандартизации (CEN) совместно с техническим комитетом по стандартизации ISO/TC 34 «Пищевая продукция» Международной организации по стандартизации (ISO) в соответствии с соглашением о техническом сотрудничестве между ISO и CEN (Венское соглашение).
Перевод с английского языка (еп).
Официальные экземпляры международного стандарта, на основе которого подготовлен настоящий государственный стандарт, и международного стандарта, на который даны ссылки, имеются в Национальном фонде ТИПА.
В разделе «Нормативные ссылки» и тексте стандарта ссылки на международный стандарт актуализированы.
Сведения о соответствии государственных стандартов ссылочным международным стандартам приведены в дополнительном приложении Д А.
Степень соответствия - идентичная (ЮТ)
4 ВЗАМЕН СТБ ISO 21571-2008
© Госстандарт, 2016
Настоящий стандарт не может быть воспроизведен, тиражирован и распространен в качестве официального издания без разрешения Госстандарта Республики Беларусь
Издан на русском языке
Приложение А
(обязательное)
Методы экстрагирования ДНК
А.1 Получение применимой для ПЦР ДНК методами экстрагирования ДНК на основе фенола/хлороформа
А.1.1 Основной метод на основе фенола/хлороформа
А.1.1.1 Общие положения
Данный метод (см. (5J) применяется для экстрагирования ДНК из широкой разновидности матриц
(см. А.1.1.8).
Фенол обычно подходит для деструкции нуклеазы и денатурации белка.
При исследовании лиственной или зеленой массы растений (например, листьев цикория, высушенной люцерны) вместе с ДНК также могут соосаждаться многие ингибиторы ПЦР. По этой причине могут возникнуть трудности, связанные с повторяемостью получения ДНК. амплифицируемой в ПЦР.
С учетом агрессивных и опасных свойств фенола в качестве альтернативы целесообразно применять методы экстрагирования ДНК. основанные на ЦТАВ. и/или поливинилпирролидоне (ПВП), и/или на адсорбции диоксидом кремния.
А.1.1.2 Статус валидации
Этот метод широко применяется во всех областях биологии, агрономии и медицины на протяжении 40 лет. однако он никогда не подвергался оценке путем межлабораторных исследований для обнаружения ГМО в пищевой продукции.
А.1.1.3 Принцип метода
Метод включает стадию лизиса (термический лизис в присутствии додецилсульфата натрия и этилендиаминтетрауксусной кислоты (EDTA) высокой концентрации) и последующее удаление загрязняющих примесей (например, липофильных молекул, полисахаридов и белков) и нуклеаз из водной фазы, содержащей ДНК, с помощью фенола и хлороформа. Заключительное осаждение этанолом концентрирует ДНК и удаляет соли и остаточный хлороформ. Критическим этапом метода является стадия лизиса [5].
А.1.1.4 Меры предосторожности
Работы с органическими реагентами необходимо выполнять в вытяжном шкафу.
А.1.1.5 Реагенты
А.1.1.5.1 Этанол, объем чистой фракции с(С?НьОН) = 96 %.
Хранить и использовать при температуре минус 20 вС.
А.1.1.5.2 Ледяная уксусная кислота (СН:,СООН).
А.1.1.5.3 Ацетат калия (С2Н302К).
А.1.1.5.4 Соляная кислота, с(НС1) = 37 %.
А.1.1.5.5 Изоамиловый спирт |(СН3)2СНСН2СН>ОН].
А.1.1.5.6 Фенол (С6НьОН).
А.1.1.5.7 Хлороформ (СНС13).
А.1.1.5.8 Трис (гидроксиметил)-аминометан (Трис) (C4H11NO3).
А.1.1.5.9 Этилендиаминтетрауксусной кислоты двукалиевая соль (K2EDTA)(CioH14N208K2).
А.1.1.5.10 Гидроксид калия (КОН).
А.1.1.5.11 Хлорид калия (KCI).
А.1.1.5.12 Додецилсульфат натрия (SDS) (C12H2t>04SNa).
А.1.1.5.13 Протеиназа-К. приблизительно 20 ед_/мг лиофилизата.
А.1.1.5.14 Рибонуклеаза-А. очищенная от ДНК. выделенная из бычьей поджелудочной железы, приблизительно 50 Kunitz ед./мг (50 000 едУмг) лиофилизата.
А.1.1.5.15 Насыщенный фенол. pH > 7.8.
Используется фенол, насыщенный буферным раствором для экстрагирования/лизиса (А.1.1.5.18), без SDS или приготовленный в соответствии с (5] или рекомендациями изготовителя.
А.1.1.5.16 Смесь хлороформ-изоамиловый спирт
Смешать 24 объемные части хлороформа (А 1.1.5.7) и одну объемную часть изоамилового спирта (А.1.1.5.5).
А.1.1.5.17 Смесь фенол-хлороформ-изоамиловый спирт
Смешать одну объемную часть насыщенного фенола (А.1.1.5.15) и одну объемную часть смеси хлороформ-изоамиловый спирт (А.1.1.5.16).
А.1.1.5.18 Буферный раствор для экстрагирования/лизиса концентрации компонентов с(Трис) = 0,050 моль/л, c(K2EDTA) = 0,050 моль/л, массовая концентрация p(SDS) = 30 г/л.
Довести pH до 8.0. используя HCI или КОН.
А.1.1.5.19 Буферный раствор ТЕ. с(Трис) = 0,010 моль/л, c(K2EDTA) = 0,001 моль/л.
Довести значение pH до 8,0, используя HCI или КОН.
А.1.1.5.20 Раствор протеиназы-К, р = 20 мг/мл, растворенный в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 °С. избегать многократного замораживания и оттаивания.
А.1.1.5.21 Раствор рибонуклеазы-А. р= 10 мг/мл лиофилизата.
Хранить при температуре минус 20 °С, избегать многократного замораживания и оттаивания.
А.1.1.5.22 Раствор этанола. с(С2Н5ОН) = 70 %.
Хранить и использовать при температуре минус 20 °С.
А.1.1.5.23 Раствор ацетата калия. с(С2Н302К) = 3 моль/л.
Довести значение pH до 5.2 ледяной уксусной кислотой. Не автоклавировать. При необходимости пропустить через фильтр с размером пор 0,22 мкм.
А.1.1.6 Оборудование
Используется обычное лабораторное оборудование, в частности следующее.
А.1.1.6.1 Центрифуга, поддерживающая ускорение не менее 10 000 д.
На некоторых стадиях анализа необходимо использование центрифуги с охлахщением.
А.1.1.6.2 Водяная баня или инкубатор с рабочим диапазоном температур от 60 °С до 70 *С.
А.1.1.6.3 Вакуумная сушилка (при необходимости).
А.1.1.6.4 Сублимационная сушилка (при необходимости).
А.1.1.6.5 Встряхиватель. например Vortex
А.1.1.6.6 Реакционные сосуды, способные выдержать заморозку в жидком азоте.
А.1.1.7 Методика
А.1.1.7.1 Общие положения
Сразу после приготовления пробы для анализа из исследуемой продукции (матрицы) необходимо выполнить описанную ниже процедуру экстрагирования и очистки ДНК. При изменении величины навески пробы требуется соответственно масштабировать массы реагентов и объемы реактивов.
А.1.1.7.2 Методика экстрагирования
Взвесить 0,25 г анализируемой пробы в микропробирку.
Добавить 1,6 мл буферного раствора для экстрагирования/лизиса (А.1.1.5.18) и, в случае необходимости (например, для проб с высоким содержанием белка). 50 мкл раствора протеиназы-К (А.1.1.5.20). Инкубировать при температуре от 60 “С до 70 °С обычно в течение от 30 мин до 2 ч (также возможно инкубирование в течение ночи). Добавить рибонуклеазу-А (А.1.1.5.21) до конечной концентрации 0,1 мкг/мл. Центрифугировать при ускорении 5000 g в течение 30 мин. Извлечь образовавшийся сверху слой (супернатант) и перенести в чистую пробирку. Добавить к нему один объем насыщенного фенола (А.1.1.5.15), затем осторожно и тщательно перемешать. Центрифугировать при ускорении 5000 g в течение 15 мин. извлечь верхнюю водную фазу и перенести в чистую пробирку. Добавить к образовавшемуся верхнему слою один объем фенола-хлороформа-изоамилового спирта (А.1.1.5.17). затем осторожно и тщательно перемешать. Центрифугировать при ускорении 5000 g в течение 15 мин, извлечь водную фазу и перенести в чистую пробирку. В зависимости от состава матрицы
^Vortex- это пример коммерчески доступного оборудования, пригодного для использования с описываемым методом. Эта информация приводится исключительно в качестве информации для пользователей настоящего стандарта и ни при каких условиях не может служить рекомендацией или одобрением конкретного типа оборудования Допускается использование аналогичного оборудования при условии, что оно позволяет получать такие же результаты
пробы данную процедуру повторяют один ипи несколько раз до тех пор. пока граница раздела фаз не станет четкой.
Добавить к образовавшемуся сверху слою один обьем хлороформа-изоамилового спирта (А. 1.1.5.16), затем осторожно и тщательно перемешать. Центрифугировать при ускорении 5000 g в течение 10 мин и извлечь верхнюю водную фазу в чистую пробирку. Повторять до тех пор. пока граница раздепа фаз не станет четкой. Смешать образовавшийся сверху слой с 0.1 объема раствора ацетата калия (А.1.1.5.23) и 2.5 объема 96%-ного этанола (А.1.1.5.1), затем тщательно перемешать, перевернув пробирки. Инкубировать в течение по меньшей мере 5 мин в жидком азоте, или 1 ч при температуре минус 80 °С. или в течение ночи при температуре минус 20 °С. Центрифугировать при ускорении 10 000 g (или вплоть до 13 000 д) при температуре 4 °С в течение по меньшей мере 15 мин, затем осторожно слить образовавшийся сверху слой.
Тщательно промыть осадок ДНК в пробирке поспе центрифугирования двумя объемами 70%-ного раствора этанола (А.1.1.5.22). Центрифугировать при ускорении от 10 000 до 13 000 g при температуре 4 °С в течение 15 мин. затем осторожно слить образовавшийся сверху слой. Эта стадия является особенно важной для удаления осаэдакнцихся солей, которые могут оказать влияние на последующий анализ (например. ПЦР).
Высушить осадок в пробирке после центрифугирования и повторно растворить его в 100 мкл воды или соответствующего буферного раствора, например буферного раствора ТЕ (А.1.1.5.19). Полученный раствор представляет собой основной раствор ДНК. Добавить рибонуклеазу-А (А.1.1.5.21) до конечной концентрации 0.1 мкг/мл.
А.1.1.8 Перечень примеров
Описанный метод был успешно применен для экстрагирования ДНК ” из следующих матриц: подкисленная соя, квашеные соевые бобы 1), обезвоженная люцерна, детское сухое печенье ”, детское молоко ”, бактерии и их споры, семена ячменя, говяжий/свиной паштет >>, пиво ”, голубой сыр, шоколадное пирожное с орехами 1), консервированная кукуруза, семена моркови, брикетированный зерновой концентрат 1>, сыр. куриные наггетсы, листья цикория, корни цикория, печенье, глазированное шоколадом 1>, шоколадная паста ,). печенье с корицей ”, компоты, кукурузные хлопья ,,1 дробленый рис, десертный крем ”, высушенные семена гороха, кукурузное печенье ”, кормовой кукурузный жмых, кукурузная мука, кукурузный глютеновый корм, семена кукурузы, гранулированный корм из маниоки. мясо в муке из маниоки, мясо свежее и подвергнутое тепловой обработке 11 (говядина, свинина. курица и индейка), мякоть дыни, семена дыни, рубленое мясо, ингредиенты мюсли 1). мюсли 1), побеги золотистой фасоли ”, семена овса, клубни картофеля, кормовой рапсовый жмых, глупаколца, семена рапса, колбаса (реализуемая в ломтиках 11 и коктейльные сосиски ” (см. А.1.2 для усовершенствованного метода экстрагирования), шницель, соус Хойсин ”, суповые шарики, соевый белок в мясной продукции ”, соевый лецитин (необработанный коричневый и желтый рафинированный 1J), побеги сои ”, соевые напитки, соя, соевый крем, кормовой соевый жмых, соевый творог, соус для спагетти ”, семена спельты, сахарная свекла (сушеный жом), сахарная свекла (свежие корнеплоды), семена сахарной свеклы, семена подсолнечника, тофу, мясистые свежие томаты, томатная паста ”, семена томатов. вегетарианский рубленый шницель, вафли (с 11 шоколадом и без ”). пшеничные отруби, пшеничная мука, глютеновый корм из пшеницы, семена пшеницы, крупка из твердой пшеницы, йогурт ” (см. А. 1.3 для усовершенствованного метода экстрагирования).
А.1.2 Метод на основе фенола-хлороформа. Процедура для заквасочных культур колбас, подвергнутых ферментации
А.1.2.1 Общие положения
Этот метод предназначен для выделения общей ДНК. включая бактериальную геномную ДНК. из колбас. Применимость метода для получения ДНК высокого качества, пригодной для специфического обнаружения рекомбинированной ДНК с использованием ПЦР. была продемонстрирована на колбасах, подвергнутых ферментации [6], а также на колбасах, подвергнутых ферментации и термической обработке, так называемых летних колбасах (7). Кроме того, было показано, что данный метод экстрагирования пригоден для выделения общей ДНК из сливок специально для обнаружения Staphylo-
” Повторяемость может зависеть от партии матрицы и/или технологии ее получения В некоторых случаях ДНК не может быть обнаружена, или она подверглась расщеплению таким образом, что результаты ПЦР ниже предела обнаружения метода независимо от используемых затравок (праймеров), протоколов ПЦР Это может быть причиной низкой воспроизводимости результатов между лабораториями
coccus aureus в этой пищевой матрице [8J. (Перечень матриц, для которых применим этот метод, приведен в А.1.2.8).
А.1.2.2 Статус валидации
Этот метод был проверен при межлабораторном исследовании на навески пробы массой 0.4 г (см. А. 1.2.9).
А.1.2.3 Принцип метода
Метод заключается в выделении бактериальных клеток из пищевой матрицы путем гомогенизации проб колбас с последующей стадией центрифугирования. Полученный осадок содержит не только бактериальные клетки, но также и частицы мяса. Для проведения специфического лизиса бактериальных клеток их оболочки расщепляют при добавлении лизоцима. Для улучшения расщепления клеточных оболочек молочнокислых бактерий мяса может быть добавлен мутанолизин. Полный лизис клеток происходит при добавлении детергента SDS (додецилсульфата натрия) и протеина-зы-К, а затем несколько раз проводится экстрагирование водной фазы фенолом и/или хлороформом. Стадия экстрагирования фенолом-хлороформом является важной для устранения любой нуклеазной активности и ингибиторов ПЦР, включая те. которые возникли из пищевой матрицы (например, гема-тин). Последним этапом является осаждение ДНК этанолом.
А.1.2.4 Меры безопасности
Работы с органическими реагентами должны выполняться в вытяжном шкафу.
А.1.2.5 Реагенты
А.1.2.5.1 Изопропанол (СН3СН(ОН)СН3].
А.1.2.5.2 Этанол, с(С?Н6ОН) = 96 %.
Хранить и использовать при температуре минус 20 °С.
А.1.2.5.3 Ледяная уксусная кислота (СН3СООН).
А.1.2.5.4 Соляная кислота. с(НС1) = 37 %.
А.1.2.5.5 Гидроксид натрия (NaOH).
А.1.2.5.6 Изоамиловый спирт ((CH^CHCH^CHpOH)).
А.1.2.5.7 Фенол (CGHbOH).
А.1.2.5.8 Хлороформ (СНС13).
А.1.2.5.9 Трис(гидроксиметил)-аминометан (Трис) (С4НпМОз).
А.1.2.5.10 Этилендиаминтетрауксусной кислоты двунатриевая соль (Na?EDTA) (CtoHuNpOeNa?).
А.1.2.5.11 Додецилсульфат натрия (SDS) (C12H?i,04SNa).
А.1.2.5.12 Лизоцим
50 ООО ед./мг белка (1 ед. будет давать Д Аад 0.001 в минуту при pH 6.24 и температуре 25 °С. используя суспензию Micrococcus lysodeikticus в качестве субстрата, в 2,6 мл реакционной смеси при оптической длине пути 1 см).
А.1.2.5.13 Сахароза (С1?Н1?0„).
А.1.2.5.14 Протеиназа-К, приблизительно 20 ед./мг лиофилизата.
А.1.2.5.15 Ацетат натрия (C^HA-Na).
А.1.2.5.16 Насыщенный фенол.
Используется фенол, насыщенный буферным раствором Трис/НС1 (pH > 7,8) или приготовленный в соответствии с [5] или рекомендациями изготовителя.
А.1.2.5.17 Смесь хлороформ-изоамиловый спирт
Смешать 24 обьемные части хлороформа (А 1.2.5.8) и одну обьемную часть изоамилового спирта (А.1.2.5.6).
А.1.2.5.18 Смесь фенол-хлороформ-изоамиловый спирт
Смешать 25 объемных частей насыщенного фенола (А.1.2.5.16) с 24 частями хлороформа (А.1.2.5.8) и одной объемной частью изоамилового спирта (А.1.2.5.6).
А.1.2.5.19 Раствор мутанолизина. содержащий 500 ед7мл или 5000 ед./мл мутанолизина. растворенного в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 °С, избегая многократного замораживания и оттаивания.
А.1.2.5.20 Раствор лизоцима, с = 10 мг/мл. растворенного в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 “С. избегая многократного замораживания и оттаивания.
А.1.2.5.21 Раствор сахарозы, с(СцНяОи) = 400 r/л.
А.1.2.5.22 Буферный раствор А, с(Трис) = 0,020 моль/л, c(Na2EDTA) = 0,020 моль/л, c(NaCI) = 0,1 моль/л.
Довести значение pH до 8,0, используя HCI или NaOH.
А.1.2.5.23 Буферный раствор для экстрагирования/лизиса, содержащий одну объемную часть буферного раствора А (А. 1.2.5.22) и одну объемную часть раствора сахарозы (А.1.2.5.21).
А.1.2.5.24 Раствор SDS, c(SDS) = 250 г/л.
А.1.2.5.25 Раствор протеиназы-К, с = 20 мг/мл.
Не автоклавировать. Хранить при температуре минус 20 вС. избегать многократного замораживания и оттаивания.
А.1.2.5.26 Раствор этанола. с(С2Н;,ОН) = 70 %.
Хранить и использовать при температуре минус 20 °С.
А.1.2.5.27 Раствор ацетата натрия, c(C2H302Na) = 3 моль/л.
Довести значение pH до 5,2 ледяной уксусной кислотой.
А.1.2.5.28 Буферный раствор ТЕ. с(Трис) = 0,010 моль/л. c(Na2EDTA) = 0,001 моль/л.
Довести значение pH до 8.0. используя HCI или NaOH.
А.1.2.6 Оборудование
Используется обычное лабораторное оборудование, в частности следующее.
А.1.2.6.1 Инструменты для измельчения пробы (например, скальпель).
А.1.2.6.2 Центрифуга, поддерживающая ускорение как минимум 12 000 д.
На некоторых стадиях анализа необходимо использовать центрифугу с охлахздением.
А.1.2.6.3 Водяная баня или инкубатор.
А.1.2.6.4 Вакуумная сушилка (при необходимости).
А.1.2.6.5 Смеситель, например Vortex 1>.
А.1.2.7 Методика
А.1.2.7.1 Общие положения
Сразу после приготовления навески пробы из исследуемой продукции (матрицы) необходимо выполнить описанную ниже процедуру экстрагирования и очистки ДНК.
При изменении величины навески пробы требуется соответственно масштабировать массу реагентов и объем реактивов.
А.1.2.7.2 Приготовление пробы
Измельчить колбасу, гомогенизировать и добавить к 200-500 мг гомогенизата три объема воды (до 1.5 мл). Выдержать при комнатной температуре приблизительно 10 мин.
А.1.2.7.3 Методика экстрагирования
Осторожно перенести 500 мкл водной фазы (суспензии) в новую пробирку. Центрифугировать в течение 10 мин при ускорении 12 000 д.
Отбросить образовавшийся сверху слой и повторно растворить осадок после центрифугирования в 500 мкл буферного раствора для экстрагирования/лизиса (А.1.2.5.23).
Добавить 58 мкл раствора лизоцима (А. 1.2.5.20). Инкубировать при температуре 37 °С в течение 1 ч. Если результаты неудовлетворительные, можно добавить к лизоциму 10 единиц мутанолизина (А. 1.2.5.19). Однако перед повседневным применением необходимо проверить специфичное для матрицы воздействие этой добавки.
Добавить 25 мкл раствора SDS (А.1.2.5.24) и 25 мкл раствора протеиназы-К (А.1.2.5.25), затем инкубировать в течение 10 мин при температуре 60 *С.
Добавить один объем фенола-хлороформа-изоамилового спирта (А.1.2.5.18) и перемешать.
Центрифугировать смесь в течение 3 мин при ускорении приблизительно 12 000 д. Перенести верхнюю водную фазу в новую пробирку.
Добавить один объем хлороформа-изоамилового спирта (А.1.2.5.17) и перемешать.
''Vortex - это пример коммерчески доступного оборудования, пригодного для использования с описываемым методом Эта информация приводится исключительно в качестве информации для пользователей настоящего стандарта и ни при каких условиях не может служить рекомендацией или одобрением конкретного типа оборудования Допускается использование аналогичного оборудования при условии, что оно позволяет получать такие же результаты
Центрифугировать смесь в течение 3 мин при ускорении приблизительно 12 ООО д. Перенести верхнюю фазу в новую пробирку.
Добавить 0.1 объема раствора ацетата натрия (А.1.2.5.27) и один обьем изопропанола (А.1.2.5.1). Осторожно перемешать несколько раз путем переворачивания пробирки.
Выдержать при комнатной температуре не менее 30 мин. Центрифугировать в течение 15 мин при ускорении приблизительно 12 ООО д. Декантировать образовавшийся сверху слой и отбросить его.
Тщательно промыть осадок после центрифугирования не менее 500 мкл раствора этанола (А. 1.2.5.26), осторожно встряхивая или перемешивая. Центрифугировать смесь в течение 10 мин при ускорении приблизительно 12 000 д. Эта стадия является особенно важной для удаления осахедаю-щихся солей, которые могут оказать влияние на последующий анализ (например, ПЦР).
Отбросить образовавшийся сверху слой.
Высушить осадок после центрифугирования и повторно растворить его в 100 мкл воды или соответствующего буферного раствора, например буферного раствора ТЕ (А.1.2.5.28). Полученный раствор представляет собой основной раствор ДНК.
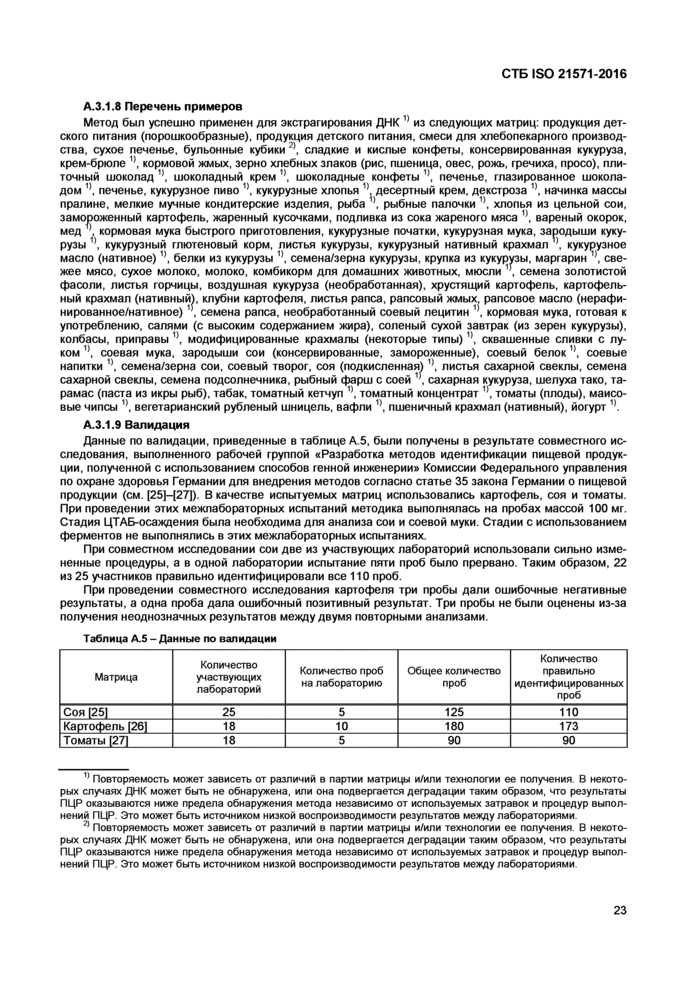
А.1.2.8 Перечень примеров
См. таблицу А.1.
|
Таблица А.1 - Перечень матриц, к которым был успешно применен данный метод | ||||||||||||
|
А.1.2.9 Валидация
Данные по валидации, приведенные в таблице А.2, были получены в результате совместного исследования, выполненного рабочей группой «Разработка методов идентификации пищевой продукции. полученной с использованием способов генной инженерии» Комиссии Федерального управления по охране здоровья Германии для внедрения методов согласно статье 35 закона Германии о пищевой продукции (6).
При проведении этого исследования две пробы дали ошибочные положительные результаты, вызванные, возможно, недостатками упаковки. Для данного исследования мутанолизин не использовался.
|
Таблица А.2 - Данные по валидации | ||||||||
|
А.1.3 Метод на основе фенола-хлороформа. Процедура для заквасочных культур йогурта
А.1.3.1 Общие положения
Этот метод описывает методику экстрагирования основной массы ДНК из заквасочных культур, используемых для ферментации йогурта из молочной продукции. Эта методика успешно применяется для обычных йогуртов, включая йогурты, содержащие различные ингредиенты, такие как фрукты, добавки и стабилизаторы, а также для продукции с различным содержанием жира (см. А.1.3.8 и [9). [10]. [11]). Из йогуртов, подвергнутых термической обработке [12], также экстрагируется ДНК, применимая для ПЦР.
А.1.3.2 Статус валидации
Этот метод был проверен при межлабораторном исследовании (см. А. 1.3.9).
A.1.3.3 Принцип метода
Этот метод основывается на методике экстрагирования фенолом-хлороформом, которая адаптирована к специальной пищевой матрице (составу) пробы. Грамм-положительные заквасочные культуры йогуртов концентрируются после расщепления коагулированного казеина при щелочном pH. Выделенные клетки повторно суспендируют в буферном водном растворе и обрабатывают лизоцимом (и мутанолизином) для расщепления клеточных оболочек. Лизис клеток происходит при добавлении ионного детергента, такого как додецилсульфат натрия (SDS). Белки удаляют путем обработки про-теиназой-К, а затем выполняют экстрагирование смесью фенол-хлороформ и хлороформом в несколько стадий. Последней стадией является осаждение ДНК этанолом.
А.1.3.4 Меры безопасности
Работы с органическими реагентами должны выполняться в вытяжном шкафу.
А.1.3.5 Реагенты
А.1.3.5.1 Изопропанол (СН3СН(ОН)СН3].
А.1.3.5.2 Этанол, с(С?НьОН) = 96 %.
Хранить и пользовать при температуре минус 20 °С.
А.1.3.5.3 Ледяная уксусная кислота (СН3СООН).
А.1.3.5.4 Хлорид натрия (NaCI).
А.1.3.5.5 Цитрат натрия (CeH;,Na30?).
А.1.3.5.6 Соляная кислота, с(НС1) = 37 %.
А.1.3.5.7 Гидроксид натрия (NaOH).
А.1.3.5.8 Изоамиловый спирт ((СН3)?СНСН?СН?(ОН)).
А.1.3.5.9 Фенол (С6Н5ОН).
А.1.3.5.10 Хлороформ (СНС1з).
А.1.3.5.11 Трис(оксиметил)-аминометан (Трис) (C4HnN03).
А.1.3.5.12 Этилендиаминтетрауксусная кислота, динатриевая соль (Na2EDTA) (C^H^NjOeNaj).
А.1.3.5.13 Додецилсульфат натрия (SDS) (C1?H^04SNa).
А.1.3.5.14 Лизоцим, 50 ООО ед./мг белка (1 ед. будет давать ЛА450 0,001 в минуту при pH 6,24 и температуре 25 °С. используя суспензию Micrococcus lysodeikticus в качестве субстрата, в 2,6 мл реакционной смеси при оптической длине пути 1 см).
А.1.3.5.15 Сахароза (С^Н^Ои).
А.1.3.5.16 Протеиназа-К, приблизительно 20 едУмг лиофилизата.
А.1.3.5.17 Ацетат натрия (C2H3C>2Na).
А.1.3.5.18 Раствор цитрата натрия. p(C(VH;,Na30/) = 400 г/л.
А.1.3.5.19 Раствор гидроксида натрия. c(NaOH) = 0,4 моль/л.
Растворить в стерильной воде. Не автоклавировать. Перед использованием готовить свежий раствор.
А.1.3.5.20 Раствор хлорида натрия/цитрата натрия (SSC 5х, концентрированный 5-кратный основной раствор), c(NaCI) = 0,75 моль/л, c(C6H6Na30/) = 0,075 моль/л.
Целесообразно готовить концентрированный основной раствор SSC 20х (например. 20-кратный основной раствор, так как растворы с высокой концентрацией солей обычно более устойчивы). Разбавлять перед использованием.
А.1.3.5.21 Насыщенный фенол
Используется фенол со значением pH 8 и насыщенный буфером Трис/НС1 (pH > 7,8) или, например, приготовленный в соответствии с [5) или рекомендациями изготовителя.
А.1.3.5.22 Смесь хлороформ-изоамиловый спирт
Смешать 24 объемные части хлороформа (А 1.3.5.10) и одну объемную часть изоамилового спирта (А.1.3.5.8).
А.1.3.5.23 Смесь фенол-хлороформ-изоамиловый спирт
Готовится путем смешивания 25 объемных частей насыщенного фенола (А. 1.3.5.21) с 24 объемными частями хлороформа (А.1.3.5.10) и одной объемной частью изоамилового спирта (А.1.3.5.8).
А.1.3.5.24 Раствор мутанолизина, содержащий 500 или 5000 едУмл муганолизина, растворенного в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 °С. избегать многократного замораживания и оттаивания.
А.1.3.5.25 Раствор лизоцима, содержащий 10 мг/мл лизоцима, растворенного в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 °С, избегать многократного замораживания и оттаивания.
А.1.3.5.26 Раствор сахарозы, ctC^H^O,,) = 400 г/л.
А.1.3.5.27 Буферный раствор А. с(Трис) = 0.020 моль/л. c(Na?EDTA) = 0,020 моль/л,
c(NaCI) = 0,100 моль/л.
Довести значение pH до 8,0 HCI или NaOH.
А.1.3.5.28 Буферный раствор для экстрагирования/лизиса, содержащий одну объемную часть буферного раствора А (А.1.3.5.27) и одну объемную часть раствора сахарозы (А.1.3.5.26).
А.1.3.5.29 Раствор SDS, p<SDS) = 250 г/л.
А.1.3.5.30 Раствор протеиназы-К. с= 20 мг/мл, растворенной в стерильной воде.
Не автоклавировать. Хранить при температуре минус 20 °С, избегать многократного замораживания и оттаивания.
А.1.3.5.31 Раствор этанола. с(С?Н:,ОН) = 70 %.
Хранить и использовать при температуре минус 20 °С.
А.1.3.5.32 Раствор ацетата натрия. с<С?Н:А>№) = 3 моль/л.
Доводят значение pH до 5,2 ледяной уксусной кислотой.
А.1.3.5.33 Буферный раствор ТЕ, с(Трис) = 0,010 моль/л, c(Na?EDTA) = 0,001 моль/л.
Довести значение pH до 8,0 HCI или NaOH.
А.1.3.6 Оборудование
Применяется обычное лабораторное оборудование, в частности следующее.
А.1.3.6.1 Центрифуга, поддерживающая ускорение не менее 12 000 д. На некоторых стадиях анализа необходимо использование центрифуги с охлаждением.
А.1.3.6.2 Водяная баня или инкубатор.
А.1.3.6.3 Вакуумная сушилка (при необходимости).
А.1.3.6.4 Смеситель, например Vortex
А.1.3.7 Методика
А.1.3.7.1 Общие положения
Сразу после приготовления навески пробы из исследуемой продукции (матрицы) необходимо выполнить описанную ниже процедуру экстрагирования и очистки ДНК. При изменении размера пробы для анализа требуется соответственно масштабировать массы реагентов и объемы реактивов.
А.1.3.7.2 Методика экстрагирования
Хорошо встряхнуть или перемешать йогурт. Перенести 250 мкл йогурта в пробирку вместимостью 2 мл. Добавить 80 мкл раствора цитрата натрия (А.1.3.5.18). Добавить 150 мкл раствора NaOH (А.1.3.5.19) и хорошо перемешать. Центрифугировать при ускорении 12 000 g в течение 2 мин.
Осадок после центрифугирования должен иметь диаметр не более 0,7 см и занимать объем не более 100 мкл. В противном случае указанные стадии (добавление 80 мкл раствора цитрата натрия и 150 мкл раствора NaOH) необходимо повторить.
Отбросить верхний слой жира и образовавшийся сверху водный слой и повторно суспендировать осадок после центрифугирования в 500 мкл раствора 5х SSC (А.1.3.5.20). Центрифугировать не менее 2 мин при ускорении около 12 000 д. Отбросить образовавшийся сверху слой. Повторно суспендировать осадок после центрифугирования в 500 мкл раствора 5х SSC. Центрифугировать в течение 2 мин при ускорении около 12 000 д. Отбросить образовавшийся сверху слой.
Повторно суспендировать осадок после центрифугирования в 500 мкл буферного раствора для экстрагирования/лизиса (А.1.3.5.28). Добавить 50 мкл раствора лизоцима (А.1.3.5.25). Инкубировать при температуре 37 °С в течение 1 ч. Если результаты неудовлетворительные, к лизоциму может быть добавлено 10 ед. мутанолизина (А.1.3.5.24). Однако перед повседневным применением необходимо проверить специфичное для матрицы воздействие этой добавки.
^Vortex- это пример коммерчески доступного оборудования, пригодного для использования с описываемым методом. Эта информация приводится исключительно в качестве информации для пользователей настоящего стандарта и ни при каких условиях не может служить рекомендацией или одобрением конкретного типа оборудования Допускается использование аналогичного оборудования при условии, что оно позволяет получать такие же результаты
Добавить 25 мкл раствора SDS (А.1.3.5.29) и 25 мкл раствора протеиназы-К (А. 1.3.5.30). Инкубировать в течение 10 мин при температуре 60 °С. Добавить 500 мкл смеси фенол-хлороформ-изоамиловый спирт (А.1.3.5.23) и перемешать. Центрифугировать в течение 3 мин при ускорении около 12 000 д. Перенести верхнюю водную фазу в новую пробирку. Добавить один обьем хлороформа-изоамилового спирта (А. 1.3.5.22) и перемешать. Центрифугировать в течение 3 мин при ускорении около 12 000 д.
Перенести верхнюю фазу в новую пробирку. Добавить 0,1 объема раствора ацетата натрия (А.1.3.5.32) и один обьем изопропанола (А.1.3.5.1). Выдержать при комнатной температуре не менее 30 мин. Центрифугировать в течение 15 мин при ускорении около 12 000 д. Отбросить образовавшийся сверху слой. Тщательно промыть осадок после центрифугирования не менее 500 мкл раствора этанола (А.1.3.5.31). Центрифугировать смесь в течение 10 мин при ускорении около 12 000 д. Эта стадия является очень важной для удаления осаждающихся солей, которые могут оказывать влияние на последующий анализ (например, ингибирование ПЦР). Отбросить образовавшийся сверху слой.
Высушить осадок после центрифугирования и повторно растворить его в 100 мкл воды или соответствующего буферного раствора, например буферного раствора ТЕ (А.1.3.5.33). Полученный раствор представляет собой основной раствор ДНК.
А.1.3.8 Перечень примеров
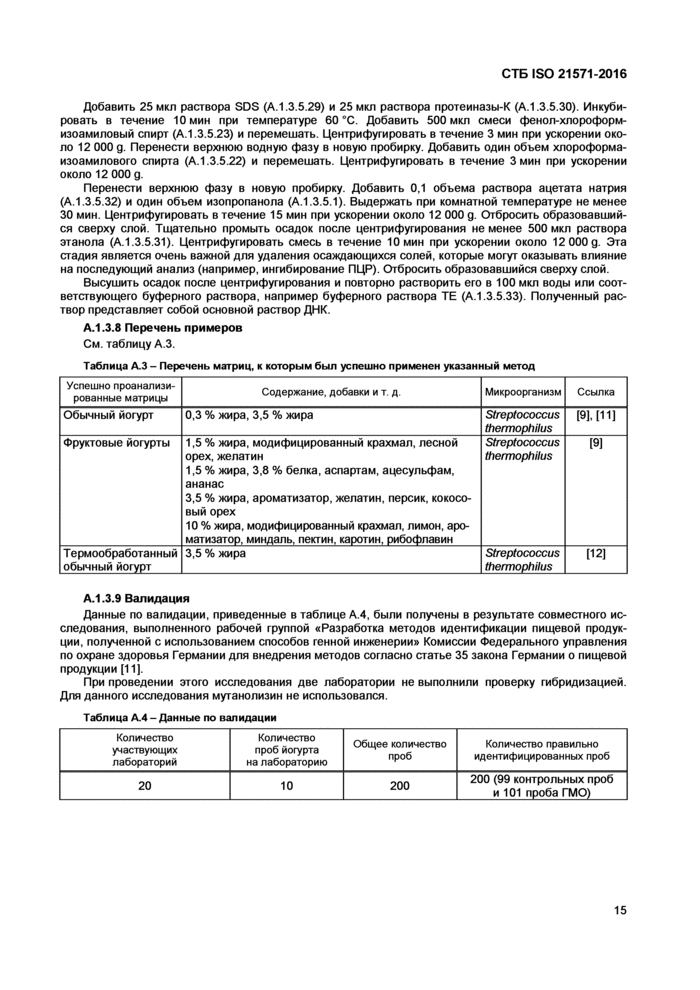
См. таблицу А.З.
|
Таблица А.З - Перечень матриц, к которым был успешно применен указанный метод | ||||||||||||||||
|
А.1.3.9 Валидация
Данные по валидации, приведенные в таблице А.4. были получены в результате совместного исследования, выполненного рабочей группой «Разработка методов идентификации пищевой продукции. полученной с использованием способов генной инженерии» Комиссии Федерального управления по охране здоровья Германии для внедрения методов согласно статье 35 закона Германии о пищевой продукции [11].
При проведении этого исследования две лаборатории не выполнили проверку гибридизацией. Для данного исследования мутанолизин не использовался.
|
Таблица А.4 - Данные по валидации | ||||||||
|
А.1.4 Метод на основе фенола-хлороформа. Процедура для дрожжей и/или гифомицетов, собранных из пищевой продукции
А.1.4.1 Общие положения
Данный метод описывает одноступенчатое экстрагирование и очистку ДНК, пригодной для ПЦР, из дрожжей, гифомицетов [13] или выделенных микробных популяций. Метод применим для экстрагирования ДНК из генетически модифицированных микроорганизмов в пробах с очень сложной матрицей [14], [15]. Метод может использоваться для экстрагирования общей ДНК из матриц [14], [15] или микробной фракции, которая непосредственно выделена из матрицы либо собрана из заквасочных культур (колонии на жидких или агаровых средах)
Примечание - Предварительное выделение микробной фракции из пробы для анализа дает наиболее достоверные результаты при экстрагировании ДНК
Общую ДНК из матрицы пробы допускается экстрагировать с помощью альтернативных методов, приведенных в приложении А. Однако эти методы не гарантируют, что достаточное количество ДНК будет выделено из всех микроорганизмов (особенно из грибов, устойчивых к лизису, или грамотрица-тельных бактерий). Данный метод может использоваться для экстрагирования общей ДНК из таких матриц, как йогурт, молоко или сыр. Однако было показано, что достоверное экстрагирование хорошо обеспечивается только для тонкоизмельченных или размолотых твердых матриц. В силу этого перед применением метода его эффективность необходимо всегда проверять на основной матрице исследуемой пробы.
А.1.4.2 Статус валидации
Этот метод экстрагирования ДНК был применен и проверен [13] на 25 родах грибов, представляющих 325 видов (включая дрожжи, используемые в хлебопекарном производстве или виноделии, и Penicillia spp. [16], используемые производителями голубого сыра), в форме мицелия и спор (среди которых виды, наиболее устойчивые к разрыву или лизису, например Aspergillus fumigatus и Cryptococcus neoformans). Данный метод был разработан таким образом, чтобы избежать лабораторного загрязнения и загрязнения между пробами. Такое свойство метода позволяет применять его для проведения повседневных экстрагирований ДНК и ПЦР в больших обьемах. При применении метода не было обнаружено изменений качества матрицы при качественной ПЦР после долгосрочного хранения при температуре минус 20 °С в течение 5 лет или между различными препаратами ДНК из одного и того же организма. Несмотря на то, что качество экстрагированной данным методом ДНК подходит для качественной ПЦР. она может подвергаться недостаточному расщеплению в процессе рестрикционного анализа. Однако для использования ДНК. экстрагированной данным методом, для количественной ПЦР необходимо выполнить процедуру ее дополнительной очистки с использованием другого метода, например такого, который описан в А.4.
А.1.4.3 Принцип метода
В основном бактерии, дрожжи или мицелий разрушаются, и ДНК одновременно экстрагируется при перемешивании с высокой скоростью в присутствиии стеклянных шариков в смеси Трис-фенол-хлороформ-ЭДТА-SDS, а затем осаждается этанолом.
А.1.4.4 Меры безопасности
Работы с органическими реагентами необходимо выполнять в вытяжном шкафу.
А.1.4.5 Реагенты
А.1.4.5.1 Этанол. cfC^OH) = 96 %.
Хранить и использовать при температуре минус 20 °С.
А.1.4.5.2 Ледяная уксусная кислота (СН:,СООН).
А.1.4.5.3 Серная кислота. c(H?S04) > 90 %.
А.1.4.5.4 Бикарбонат калия (КНСОз).
А.1.4.5.5 Ацетат калия (C2H3O2K).
А.1.4.5.6 Соляная кислота. с(НС1) = 37 %.
А.1.4.5.7 Изоамиловый спирт [(СНзЬСНСНгСНгОН].
А.1.4.5.8 Фенол (С6Нг,ОН).
А.1.4.5.9 Хлороформ (СНС13).
А.1.4.5.10 Трис(оксиметил)-аминометан (Трис) (СдНцМОз).
А.1.4.5.11 Этилендиаминтетрауксусная кислота, дикалиевая соль (K?EDTA) (CioH^NjOeKp).
А.1.4.5.12 Гидроксид калия (КОН).
Содержание
1 Область применения......................................................
2 Нормативные ссылки......................................................
3 Принцип метода..............................................................
3.1 Общие положения....................................................
3.2 Экстрагирование ДНК..............................................
3.3 Количественная оценка содержания ДНК.....................................................................................2
4 Общие требования к лаборатории........................................................................................................2
5 Методика.................................................................................................................................................2
5.1 Приготовление пробы для анализа................................................................................................2
5.2 Экстрагирование и очистка ДНК.....................................................................................................3
5.3 Количественная оценка экстрагированной ДНК...........................................................................5
5.4 Стабильность экстрагированной ДНК............................................................................................6
6 Оценка результатов................................................................................................................................6
7 Протокол испытаний...............................................................................................................................6
Приложение А (обязательное) Методы экстрагирования ДНК............................................................7
Приложение В (обязательное) Методы количественной оценки экстрагированной ДНК................36
Библиография..........................................................................................................................................42
Приложение Д А (справочное) Сведения о соответствии государственных стандартов
ссылочным международным стандартам........................................................................47
А.1.4.5.13 Додецилсульфат натрия (SDS) (Ci7H»04SNa).
A.1.4.5.14 Рибонуклеаза-А, свободная от дезоксирибонуклеазы, выделенная из бычьей поджелудочной железы, приблизительно 50 Kunitz ед./мг лиофилизата.
А. 1.4.5.15 Насыщенный фенол. pH > 7,8.
Используют фенол (А.1.4.5.8), приготовленный в соответствии с (5] или в соответствии с рекомендациями изготовителя или (по выбору) полностью насыщенный буферным раствором для экстрагирования (А.1.4.5.18) без SDS.
А.1.4.5.16 Смесь хлороформ-изоамиловый спирт
Смешать 24 объемные части хлороформа (А.1.4.5.9) с одной объемной частью изоамилового спирта (А.1.4.5.7).
А.1.4.5.17 Смесь фенол-хлороформ-изоамиловый спирт
Смешать одну объемную часть насыщенного фенола (А.1.4.5.15) с одной объемной частью хло-роформа-изоамилового спирта (А.1.4.5.16).
А.1.4.5.18 Буферный раствор для экстрагирования/лизиса, с(Трис) = 0.050 моль/л,
c(K?EDTA) = 0,050 моль/л. c(SDS) = 30 г/л.
Довести значение pH до 8.0 HCI или КОН.
А.1.4.5.19 Буферный раствор ТЕ, с(Трис) = 0,010 моль/л, c(K?EDTA) = 0,001 моль/л.
Довести значение pH до 8.0 HCI или КОН.
А.1.4.5.20 Раствор рибонуклеазы-А. pfRNase-A) = 10 мг/мл лиофилизата.
Хранить при температуре минус 20 °С.
А.1.4.5.21 Раствор этанола, с(С?Н<,ОН) = 70 %.
Хранить и использовать при температуре минус 20 *С.
А.1.4.5.22 Раствор ацетата калия, с(С2НзО?К) = 3 моль/л.
Довести значение pH до 5.2 ледяной уксусной кислотой. Не автоклавировать. При необходимости пропускают через фильтр с размером пор 0,22 мкм.
А.1.4.5.23 Кондиционированные стеклянные шарики
Стеклянные шарики диаметром 0,2-0,5 мм выдержать в течение ночи в концентрированной H?S04 (А.1.4.5.3). Промыть их стерильной водой, прокипятить в растворе КНСОз (А.1.4.5.24), снова промыть стерильной водой и высушить при температуре 80 °С в вакууме (13), (14).
А.1.4.5.24 Раствор бикарбоната калия. р(КНСОз) = 50 г/л.
Использовать свежеприготовленный водный раствор.
А.1.4.6 Оборудование
Используется обычное лабораторное оборудование, в частности следующее.
А.1.4.6.1 Встряхиватель для полиэтиленовых микропробирок вместимостью 2 мл с навинчивающимися колпачками. Скорость встряхивания- не менее 100 встряхиваиий/мин (например, Mini-BeadBeaterTM 1J).
А.1.4.6.2 Микропробирки, полиэтиленовые пробирки вместимостью 2 мл с кольцевым уплотнением и навинчивающимися колпачками.
А.1.4.6.3 Устройство для фильтрования, фильтры из стекловолокна диаметром 25 мм.
А.1.4.6.4 Центрифуга, способная удерживать микропробирки вместимостью 2 мл при ускорении как минимум 10 000 д.
На некоторых стадиях анализа необходимо использование центрифуги с охлаждением.
А.1.4.6.5 Водяная баня или инкубатор.
А.1.4.6.6 Вакуумная сушилка (при необходимости).
Рекомендуется для приготовления стеклянных шариков (А.1.4.5.23).
А.1.4.6.7 Смеситель, например Vortex ?).
" Mim-BeadBeater - пример подходящего оборудования, имеющегося в продаже из Biospec-оборудования Эта информация приводится для удобства пользователей настоящего стандарта и не предполагает неукоснительное применение Может использоваться эквивалентное по параметрам оборудование, если его применение приводит к тем же результатам
21 Vortex - это пример коммерчески доступного оборудования, пригодного для использования с описываемым методом Эта информация приводится исключительно в качестве информации для пользователей настоящего стандарта и ни при каких условиях не может служить рекомендацией или одобрением конкретного типа оборудования Допускается использование аналогичного оборудования при условии, что оно позволяет получать такие же результаты
Введение
Исследование генетически модифицированных организмов (далее - ГМО) и производных продуктов осуществляется посредством следующих стадий, выполняемых последовательно или одновременно. После отбора проб нуклеиновые кислоты экстрагируются из навески пробы. Экстрагированные нуклеиновые кислоты далее могут быть очищены в процессе экстрагирования или после него. Следующими этапами являются: оценка количества экстрагированных нуклеиновых кислот (при необходимости), разведение нуклеиновых кислот (при необходимости) и выполнение аналитических процедур, например полимеразной цепной реакции (далее - ПЦР). Указанные этапы подробно изложены в настоящем стандарте и следующих международных стандартах:
- ISO 21569 «Продукты пищевые. Методы анализа на обнаружение генетически модифицированных организмов и их производных продуктов. Качественные методы, основанные на нуклеиновой кислоте»;
- ISO 21570 «Продукты пищевые. Методы анализа на обнаружение генетически модифицированных организмов и их производных продуктов. Количественные методы, основанные на нуклеиновой кислоте».
Дополнительная информация об общих требованиях и определениях, относящихся к упомянутым выше этапам, приведена в ISO 24276 «Продукты пищевые. Методы анализа для обнаружения генетически модифицированных организмов и полученных из них продуктов. Общие требования и определения».
_ГОСУДАРСТВЕННЫЙ СТАНДАРТ РЕСПУБЛИКИ БЕЛАРУСЬ_
ПРОДУКЦИЯ ПИЩЕВАЯ Методы анализа для обнаружения генетически модифицированных организмов
и производных продуктов Экстрагирование нуклеиновых кислот
ПРАДУКЦЫЯ ХАРЧОВАЯ Метады анал1зу для выяулення генетычна мадыфшаваных аргажзмау
i вытворных прадуктау Экстрагаванне нуклешавых юслот
Foodstuffs
Methods of analysis for the detection of genetically modified organisms and derived products
Nucleic acid extraction
Дата введения 20174)1-01
1 Область применения
Настоящий стандарт устанавливает общие требования и специфические методы экстрагирования. очистки и количественной оценки дезоксирибонуклеиновой кислоты (далее - ДНК). Эти методы изложены в приложениях А и В.
Настоящий стандарт разработан для пищевых матриц, но может быть также применен к другим матрицам, например зерновым культурам и кормам.
Настоящий стандарт следует применять совместно с ISO 21569, ISO 21570 в части аналитических методов на основе нуклеиновых кислот, в частности качественных аналитических методов, установленных в ISO 21569. и количественных аналитических методов, установленных в ISO 21570.
2 Нормативные ссылки
Для применения настоящего стандарта необходим следующий ссылочный стандарт. Для датированных ссылок применяют только указанное издание ссылочного стандарта, для недатированных ссылок применяют последнее издание ссылочного стандарта (включая все его изменения).
ISO 24276:2006 Продукты пищевые. Методы анализа для определения генетически модифицированных организмов и полученных из них продуктов. Общие требования и определения
3 Принцип метода
3.1 Общие положения
Основной целью применения методов экстрагирования нуклеиновых кислот является обеспечение выделения нуклеиновых кислот, пригодных для последующего анализа (см. ISO 24276).
Примечание - «Качество» ДНК зависит от средней длины экстрагированных молекул ДНК. химической чистоты и структурной целостности одной нити и двойной спирали ДНК (например, внутри- и межцелочечные связи оснований ДНК. выпадение оснований в цепочке ДНК. перекрестные сшивки с высокомолекулярными спиртами, гемином и т. д.). Кроме того, такие структурные изменения часто являются специфичными для определенной последовательности ДНК и поэтому не распределены по геному случайным образом (см (1}-[4])
Пользователям настоящего стандарта необходимо иметь в виду, что некоторые методы (например. методы на основе диоксида кремния) могут быть запатентованы.
3.2 Экстрагирование ДНК
Основной принцип экстрагирования ДНК заключается в выделении ДНК. присутствующей в матрице пробы, а затем одновременной или последующей очистке ДНК от ингибиторов ПЦР.
Издание официальное
Методы экстрагирования и очистки ДНК изложены в приложении А. Выбор метода основывается на опыте пользователя с учетом области применения и примеров матриц, которые указаны в каждом методе.
Альтернативные методы могут применяться при условии, что метод был проверен на соответствующей матрице, подлежащей исследованию.
3.3 Количественная оценка содержания ДНК
Количественная оценка экстрагированной ДНК может потребоваться для последующего анализа ПЦР.
Такая оценка может выполняться либо физическими (например, измерением поглощения на специфической длине волны), физико-химическими (например, с применением интеркалирующих или связующих агентов, обладающих флюоресцентными свойствами), ферментативными (например, биолюмииесцентным обнаружением) методами, либо путем количественной ПЦР. Последний метод применяется для составных и сложных матриц, проб с низким содержанием ДНК или проб, в которых ДНК подверглась деградации.
Существует несколько методов, применяемых для определения количества ДНК, присутствующей в растворе. Эти методы изложены в приложении В. Выбор наиболее подходящего метода осуществляется пользователем в зависимости от количества и качества ДНК, подлежащей количественному определению, а также от матрицы, из которой была экстрагирована ДНК.
Альтернативные методы могут применяться при условии, что метод был проверен на соответствующей матрице.
4 Общие требования к лаборатории
Вследствие воздействия пыли и распыляемых аэрозолей может происходить случайная контаминация ДНК. Вследствие этого организация рабочих мест в лаборатории основывается на строгом соблюдении:
- всех установленных методологических этапов, используемых для получения промежуточных и конечных результатов; и
- принципа «прямого потока» при обращении с пробами.
Использование последнего принципа гарантирует, что ДНК, подлежащая анализу, и амплифици-рованная ДНК, полученная в результате ПЦР, остаются физически разделенными.
Дополнительные требования к лабораториям приведены в ISO 24276.
5 Методика
5.1 Приготовление пробы для анализа
5.1.1 Общие положения
Специфические параметры анализируемой продукции (например, влажность) и ее обработка могут влиять на количество и качество ДНК, экстрагируемой из анализируемой продукции. В связи с этим рабочие характеристики применяемого метода экстрагирования ДНК зависят от матрицы анализируемой продукции.
Необходимо принять соответствующие меры, чтобы гарантировать, что навеска пробы является представительной лабораторной пробой.
Навеска пробы должна быть достаточного размера (объема, массы) и содержать достаточное количество частиц для обеспечения представительности лабораторной пробы.
Исходя из практических и технических соображений, не рекомендуется, чтобы величина навески пробы превышала 2 г.
Любые ограничения, связанные с размером навески пробы, не позволяющие рассматривать ее как представительную пробу, должны быть учтены при интерпретации аналитических результатов и указаны в протоколе испытаний. Методы экстрагирования ДНК, приведенные в приложении А, устанавливают величину навески пробы от 200 до 500 мг, что обычно является приемлемым для продукции с высоким содержанием ДНК (твердое и дробленое зерно, мука). Однако для матриц некоторой продукции, содержащих очень малые количества ДНК или деградировавшую ДНК. указанная навеска пробы не позволяет экстрагировать достаточное для анализа количество ДНК. В таких случаях размер навески пробы может быть увеличен.
Экстрагирование ДНК должно выполняться не менее чем из двух навесок пробы одной и той же продукции.
Хранение стандартных образцов, лабораторных проб и навесок проб должно соответствовать требованиям ISO 24276 и быть организовано таким образом, чтобы анализируемые биохимические параметры были сохранены (подробнее см. ISO/IEC 17025).
5.1.2 Пробы
Все операции по приготовлению лабораторных проб (например, измельчение, гомогенизация, деление, высушивание) должны выполняться в соответствии с лабораторными схемами, описанными в ISO 24276:2006 (пункт 5.3.2). При выполнении всех необходимых операций должны быть приняты меры предосторожности для защиты лабораторной пробы от загрязнения или изменения ее состава.
Лабораторные пробы должны быть достаточно однородными перед их сокращением и измельчением. перед отбором анализируемой части пробы.
В случае приготовления жидких проб необходимо встряхивать сосуд с пробой для обеспечения гомогенизации продукции. В случае негомогенной продукции типа неочищенных масел необходимо убедиться, что осадок и все нерастворимые составляющие полностью удалены со стенок сосуда.
В случае приготовления проб с твердыми матрицами, отличающимися затрудненным измельчением, проба должна быть измельчена для уменьшения размера частиц и/или улучшения экстрагиру-емости ДНК. В таких случаях особое внимание необходимо обратить на размер частиц. Навеска пробы, подвергаемая экстрагированию, должна содержать минимальное количество частиц. Необходимо, чтобы оборудование, предназначенное для дробления и измельчения пробы, было легко моющимся по всей рабочей поверхности и обеспечивало требуемое количество и размер частиц (гранулометрический состав) анализируемой пробы.
Если какие-либо компоненты лабораторной пробы были удалены до проведения экстрагирования. то это должно быть указано в протоколе испытаний с описанием проведенных операций.
Для твердой или пастообразной готовой пищевой продукции с высоким содержанием липидов достаточно трудно достичь требуемого размера частиц за один этап измельчения. В таких случаях допускается перед измельчением применять дополнительные процедуры, такие как удаление липидов гексаном после промежуточного измельчения, замораживание или сублимационная сушилка.
Для улучшения измельчения пастообразной или вязкой продукции для некоторых матриц можно использовать одну из следующих обработок:
- нагрев до максимальной температуры 40 °С;
- растворение в соответствующей жидкости, например воде;
- замораживание при температуре минус 20 °С или ниже.
После такой обработки следует гомогенизировать всю лабораторную пробу. Затем необходимо отобрать две навески пробы с учетом возможного проведения в дальнейшем процедур разбавления или концентрирования.
Во время проведения процедур размола и измельчения необходимо соблюдать меры, гарантирующие поддержание нагрева лабораторной пробы на минимальном уровне и не допускающие ее сильного нагревания, так как нагрев может отрицательно воздействовать на качество экстрагируемой ДНК.
По возможности необходимо избегать способов размола и измельчения, при которых возникает высокий риск перекрестного загрязнения (например, объединенное использование жидкого азота и ступки, а также использование одной ступки для разных проб). Одним из основных требований является необходимость изолирования любого методологического этапа, производящего пыль, от всех других аналитических этапов и процедур.
В случае присутствия в лабораторной пробе соли, пряностей, сахарной пудры, сахара-песка, специй и/или других веществ, которые могут потенциально оказывать влияние на экстрагирование или дальнейший аналитический этап, должны быть предприняты дополнительные соответствующие процедуры очистки в соответствии с используемым методом (см. приложение А).
Например, в случае приготовления проб сложного состава материал для исследования (матрица) (например, панировочный слой рыбных палочек) может быть отделен от остальной части продукции для экстрагирования ДНК.
5.2 Экстрагирование и очистка ДНК
5.2.1 Общие положения
При разработке методов экстрагирования необходимо учитывать следующие требования.
Качество и количество экстрагируемой нуклеиновой кислоты при использовании заданного метода на заданной матрице пробы должны быть как повторяемыми, так и воспроизводимыми при условии содержания достаточного количества нуклеиновой кислоты в матрице пробы, из которой она была экстрагирована.
Для получения ДНК хорошего качества рекомендуется при необходимости удалить следующие компоненты:
- полисахариды (пектин, целлюлозу, гемицеллюлозу, крахмал, загустители и т. д.) путем обработки соответствующим ферментом (например, пектиназой, целлюлазой, гемицеллюлазой, а-амилазой) или экстрагирования органическим растворителем (например, гексадецилтриметилам-монийбромид (ЦТАБ)/хлороформ);
-рибонуклеиновую кислоту (далее- РНК) и/или белки путем соответствующей обработки, например ферментативной обработкой рибонуклеазой и протеиназой соответственно;
- липидные фракции путем, например, ферментативной обработки или использования растворителей (например, н-гексана);
- соли (например, из буферного раствора для экстрагирования и лизиса на стадии осаждения), способные оказывать влияние на последующий анализ.
В частности, в случае приготовления твердых или высушенных проб объем буферного раствора для экстрагирования и лизиса должен быть таким, чтобы гарантировать растворение в нем ДНК.
Примечание 1 - Очистка ДНК может выполняться различными способами, например путем фракционного осаждения с использованием таких растворителей, как фенол, хлороформ, этанол, изопропанол, и/или с использованием адсорбции на твердые матрицы (аниомообменную смолу, силикагель или стеклянный гель, диатомовую землю, мембраны ит. д ). Можно объединить несколько принципов очистки ДНК При необходимости экстрагирование и очистка могут совмещаться путем выполнения на одном и том же этапе
В случае использования соосадителей ДНК. например гликогена, полиэтиленгликоля (ПЭГ), транспортной РНК (т-РНК), для увеличения соосажаения ДНК они не должны обладать нуклеазной активностью (содержать регистрируемый уровень нухлеаз), содержать ингибиторы и/или конкуренты ПЦР или последовательности нуклеиновых кислот, гомологичные анализируемым последовательностям. При выделении ДНК из генетически модифицированных растений в качестве соосадителя может быть использована, например. ДНК спермы лосося или сельди.
При использовании сублимационных сушилок для высушивания осадка ДНК, полученного после стадии осаждения, необходимо учитывать риск перекрестного загрязнения.
Необходимо повторно растворить ДНК в воде или буферном растворе для предотвращения деградации ДНК.
Пример - Буферный раствор Трис/EDTA (этилендиаминтетрауксусная кислота, EDTA) (буферный раствор ТЕ, 1* или 0,1*) с установленным значением pH 8,0, который используется для перемешивания или разбавления ДНК.
При применении нового метода экстрагирования ДНК или применении одного из методов, описанных в приложении А. для новых объектов потенциальные качество и целостность экстрагированной ДНК с использованием выбранного метода должны быть проверены следующим образом: добавлением в лизирующий буферный раствор известного количества меченой ДНК вместе с объектом, из которого необходимо выделить ДНК. Если оценка нового метода производится на основании использования меченой ДНК, количество (масса) которой известно, или если оценка производится на основе подсчета количества копий определенной последовательности ДНК, необходимо оценить возможную перекрестную реактивность такой ДНК с ДНК. содержащейся в исследуемой матрице.
Примечание 2 - Использование меченой ДНК является наилучшим приближением к реальной ситуации, когда предполагается образование комплекса ДНК данной матрицы с другими компонентами (например, белками) Такой метод также может использоваться для оценки присутствия растворимых и трансдействующих ингибиторов ПЦР в экстрагированной ДНК (см ISO 24276, ISO 21569 и ISO 21570)
Однако меченая ДНК может привести к ложному представлению о степени извлечения ДНК, так как она может гораздо легче отделяться от матрицы, чем ДНК-мишень.
5.2.2 Процедуры контроля
Процедуры контроля, подлежащие использованию при проведении анализа, описаны в таблице 1 ISO 24276. Они как минимум должны включать отрицательный и положительный контроль экстрагирования, а также контроль помещений и окружающей среды.
5.2.3 Контроль чистоты ДНК. Внутренний контроль ПЦР
При отработке нового метода экстрагирования присутствие ингибиторов ПЦР в экстрагированной ДНК может быть оценено с помощью внесения ДНК (см. ISO 24276, ISO 21569 и ISO 21570). Количество добавленной ДНК не должно превышать максимального уровня, допустимого для применяемой системы ПЦР, и должно содержать установленное число копий последовательности мишени. Это число необходимо определять отдельно для каждой последовательности мишени и указывать как кратное нижнему пределу обнаружения. В идеальном случае концентрация мишени в ПЦР при позитивном контроле должна соответствовать чувствительности, требуемой для анализа. При использовании в качестве положительного контроля ПЦР высококонцентрироваиной клонированной искомой последовательности ДНК необходимо соблюдать осторожность из-за высокой опасности перекрестного загрязнения. По возможности нуклеиновые кислоты положительных контролей должны максимально соответствовать параметрам нуклеиновых кислот исследуемого материала.
5.3 Количественная оценка экстрагированной ДНК
5.3.1 Общие положения
Качество, целостность и количество матрицы нуклеиновой кислоты оказывают влияние на рабочие характеристики аналитического метода и, следовательно, на полученные аналитические результаты. Предел обнаружения конкретного метода может, таким образом, зависеть от количества использованных нуклеиновых кислот. Общее количество ДНК, используемой в ПЦР, должно определяться вместе с общим количеством целевой таксон-слецифичной ДНК, которое должно учитываться, поскольку нецелевая таксон-специфичная ДНК может оказывать отрицательное влияние на эффективность ПЦР.
Количественная оценка ДНК помогает при:
- определении эффективности различных протоколов выделения ДНК для определенного вида объектов (повторяемость); и
- измерении концентрации нуклеиновых кислот перед анализом.
5.3.2 Область применения
Каждый метод количественной оценки должен применяться в пределах его динамического диапазона с учетом уровня прецизионности.
5.3.3 Количественные стандартные образцы
Точность методов количественной оценки зависит от стандартных образцов нуклеиновых кислот, используемых для калибровки метода.
При использовании метода, чувствительного к размеру и/или качеству фрагментов нуклеиновой кислоты, должны применяться стандартные образцы нуклеиновой кислоты, которые соответствуют размеру и/или качеству ожидаемой нуклеиновой кислоты, экстрагированной из пробы.
Используемый референсный материал (национальный или международный стандартный образец) должен обеспечивать прослеживаемость заявленных характеристик по непрерывной цепи сравнения до национального или международного эталона [см. ISO Guide 30].
При применении метода с использованием интеркапирующих агентов необходимо применять стандартный образец ДНК с высокой молекулярной массой, если количественной оценке подлежит ДНК с высокой молекулярной массой. Соответственно, необходимо использовать стандартный образец ДНК с низкой молекулярной массой при количественной оценке ДНК с низкой молекулярной массой. Нуклеиновая кислота с высокой молекулярной массой, как правило, содержит некоторое количество фрагментов с более низкой молекулярной массой. Это означает, что многие методы количественной оценки ДНК имеют определенную степень неточности, которую необходимо учитывать при оценке результатов.
Примечание - В зависимости от исследуемого материала и применяемого метода экстрагирования некоторая часть экстрагируемой ДНК может быть извлечена в виде одноцепочечной ДНК (которая имеет гораздо
более низкую способность к интеркаляции), что приводит к занижению общего содержания ДНК С другой
стороны, одноцепочечная ДНК также хорошо обнаруживается путем физических измерений
Для построения калибровочного графика требуется не менее трех точек, предпочтительно в повторах. Количество стандартного образца ДНК, используемого для каждой точки калибровочного графика, зависит от чувствительности метода и динамического диапазона измерений.
5.4 Стабильность экстрагированной ДНК
Экстрагированная ДНК должна храниться в условиях, которые обеспечивают ее стабильность для проведения последующих анализов.
Следует избегать многократного замораживания и оттаивания растворов ДНК.
6 Оценка результатов
Применяемый метод экстрагирования ДНК должен обеспечивать получение нуклеиновой кислоты, качество и количество которой соответствуют требованиям последующих анализов.
Качество выделенных нуклеиновых кислот должно быть удостоверено соответствующим аналитическим методом с регистрацией и оценкой параметров, аналогичных используемым при анализе (например, если выполняемым анализом является ПЦР, качество экстрагированной ДНК должно оцениваться с использованием соответствующих средств контроля ПЦР).
Дополнительные требования для оценки совместимости метода указаны в ISO 21569. ISO 21570 и ISO 24276.
7 Протокол испытаний
В случае представления протокола испытаний в соответствии с ISO 24276 в него должна быть включена следующая дополнительная информация о деятельности лаборатории:
- описание происхождения проб для анализа и предварительной обработки пробы перед экстрагированием нуклеиновой кислоты;
- размер проб для анализа, используемых для экстрагирования нуклеиновой кислоты;
- используемый метод экстрагирования нуклеиновой кислоты;
- любые нетипичные ситуации, наблюдаемые во время проведения испытаний;
- любая операция, не указанная в методе или рассматриваемая как необязательная, которая может оказать влияние на результаты;
- оценка результатов;
- имя оператора.
Обработка и хранение исходных данных изложены в ISO/IEC 17025 и определяются системой менеджмента качества. Необходимо обеспечить соответствие требованиям, установленным в указанных документах.